南湖新闻网讯(通讯员 叶青 )11月11日,农业微生物资源发掘与利用全国重点实验室、湖北洪山实验室、生命科学技术学院益生菌智造创新团队彭楠教授课题组在Cell Discovery期刊在线发表题为“Reprogramming an RNA-guided archaeal TnpB endonuclease for genome editing”的研究论文。该研究开发了全球首个来自生命三域之一古菌域的RNA引导的微型编程性核酸酶系统(SisTnpB1),在多种不同生长温度的微生物中实现高效基因编辑。该成果有望突破 “卡脖子”技术难题,为现代生物育种提供自主知识产权的高效基因编辑工具。
基于CRISPR-Cas9或Cas12a的基因编辑工具已经得到广泛的应用,成为推动生命科学、现代农业、医学和合成生物学等领域快速发展的重要动力。但是,基因编辑工具的应用仍面临巨大挑战。例如, 由于Cas9及Cas12a核酸酶系统分子量巨大(通常大于1000个氨基酸),限制了递送效率等。更重要的是,由于缺乏CRISPR-Cas9及Cas12a的底层知识产权,基于该系统的基因编辑技术成为我国现代生物育种的“卡脖子”技术难题。
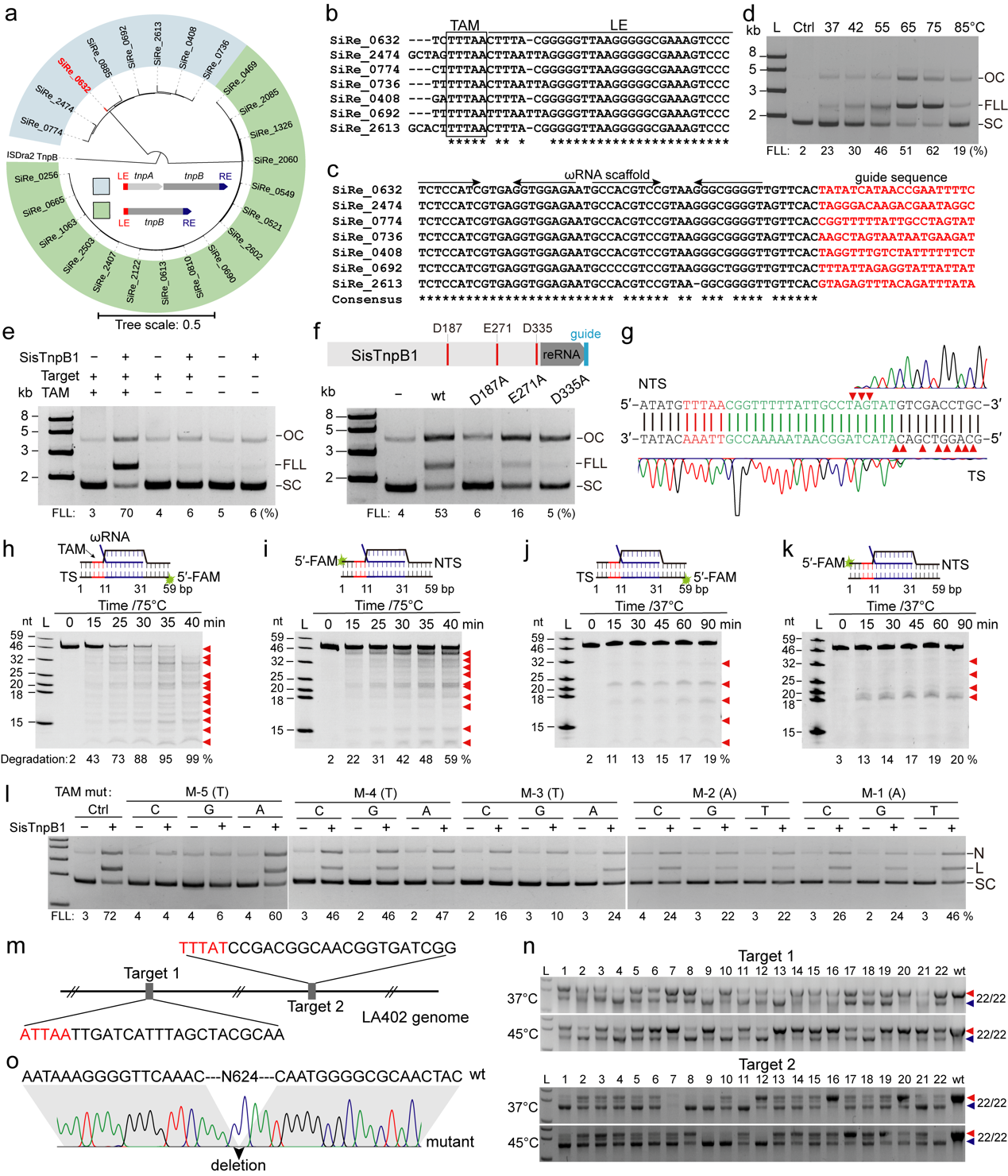
图1. 鉴定全球首个来自古菌的微型编程酶性核酸酶系统SisTnpB1并开发为高效的基因编辑工具
目前,所有Cas9及Cas12a等编程性核酸酶均来自细菌。而古菌编码的编程酶没有得到有效的发掘。农业微生物资源发掘与利用全国重点实验室、湖北洪山实验室、生命科学技术学院益生菌智造创新团队彭楠教授课题组从古菌Sulfolobus islandicus REY15A中鉴定了23个IS200/605家族编码的TnpB编程酶性核酸酶。该酶具有分子量小(40 kDa)、与已报道的RNA引导的编程性核酸酶同源性低(<30%)等特点,具有开发成自主知识产权基因编辑工具的巨大潜力。该研究团队通过生信分析,准确预测了引导RNA结构和识别靶标DNA的序列以及引发TnpB切割的TAM(Transposon-Associated Motif)序列。通过纯化其中一个典型的TnpB(命名为SisTnpB1)及其结合的引导RNA核糖核蛋白复合物(RNP)进行体外核酸切割实验,发现SisTnpB1在较广的温度活性范围内(37 ~ 85℃)均具有活性,其中最适温度在65 ~ 75℃之间。此外,SisTnpB1具有TAM依赖的切割特异性并且是Mg2+和Mn2+依赖特性。通过测序分析切割产物,发现其与Cas12家族蛋白具有相似的DNA交错切割特点,突变SisTnpB1的RuvC域的任意保守氨基酸均严重降低其切割活性。通过对靶标DNA碱基突变分析,表明靶标序列最关键的“种子”序列位于+1到+10的位置,且靶标序列+1核苷酸突变强烈抑制SisTnpB1 RNP的切割活性。此外,SisTnpB1只需一个不保守的TAM序列(5 '-WNHNN-3 ')即可用于引发靶标DNA的切割。
该团队在细菌体内验证了SisTnpB1的靶向切割活性。通过设计靶向质粒等外源遗传元件的引导RNA,SisTnpB1系统可以在细菌体内37℃条件下实现特异性切割并消除这些遗传元件。最终,该团队将SisTnpB1系统开发成为全球首个来自古菌的微型基因编辑工具,在乳酸片球菌(37和45℃)及冰岛硫化叶菌(75℃)中实现了精确的基因编辑,编辑效率>90%。上述成果近期发表在Cell Discovery期刊上,并申请中国发明专利和PCT专利各一项。
生科院徐颖博士研究生为论文第一作者,刘涛博士后为论文第二作者,彭楠教授为论文通讯作者。生科院硕士研究生王静、熊彬杨以及已经毕业的刘玲博士对该工作提供了帮助。农业微生物资源发掘与利用全国重点实验室为该成果提供了科研平台。该研究得到了国家重点研发计划、国家自然科学基金和湖北洪山实验室重大项目资助。
原文链接:https://www.nature.com/articles/s41421-023-00615-2
通讯员:叶青
